产品介绍
CLC微生物基因组学模块能够使微生物学专家充分利用全基因组或者宏基因组的数据。分析比较微生物群落的分类和功能组成,或使用病原体的分类功能来确认病原体的身份和特征。研究人员可以在流行病学样本信息的背景下来研究outbreak trees,其中病原体信息,数据和结果都可以通过一个方便的Outbreak指数来进行管理。
产品特色
微生物分析
1、微生物群落的分类学分析
(1)扩增子测序数据
1) 有无参考基因组的16S-,18S-,及其ITS rRNA数据的OTU聚类。
2) 支持通用分类法数据库Greengenes、Silva和UNITE及其定制的数据库。
(2)鸟枪宏基因组数据
1) 对宏基因组样本中的微生物的测定具有比较高的精度和相对丰度。
2) 过滤掉来自宿主DNA的污染数据。
3) 希望使用CosmosID高度管理的参考数据库的用户可以使用CosmosID插件进行微生物组分析。
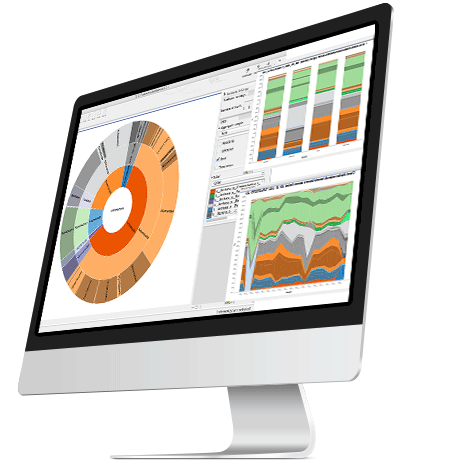
2、微生物群落的功能性分析
1) 以一流的精度和性能组装注释宏基因组的元数据。
2) 使用FAM、基因本体论和BLAST软件进行功能分析。
3) 宏基因组的功能注释需要 MetaGeneMark插件。
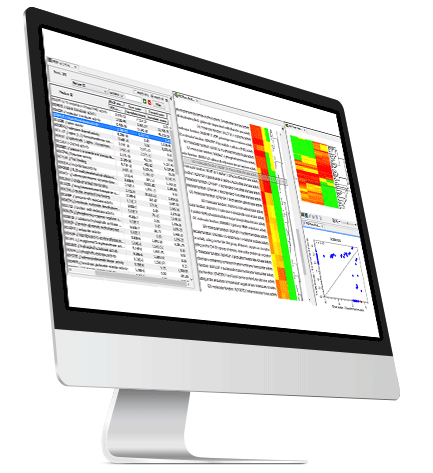
元数据的可视化和统计分析
1) 用于Alpha和Beta多样性评估的用户友好的多样性分析工具,用于比较样本间的多样性和组成相似性。
2) 基于微生物组成的2D和3D的主坐标分析(PCoA)图以及样本聚类热图。
3) 运行PERMANOVA分析,评估对聚类的统计。
4) 合并丰度表,以便在样本间直接比较多样性指标。
5) 探索不同样本类别间差异最大的分类单元或基因功能。利用最先进的多因素差分丰度分析方法,测量折线变化,估计统计意义,探讨样本类别间的相关性。
6) 利用样本元数据进行绘图和统计测试。
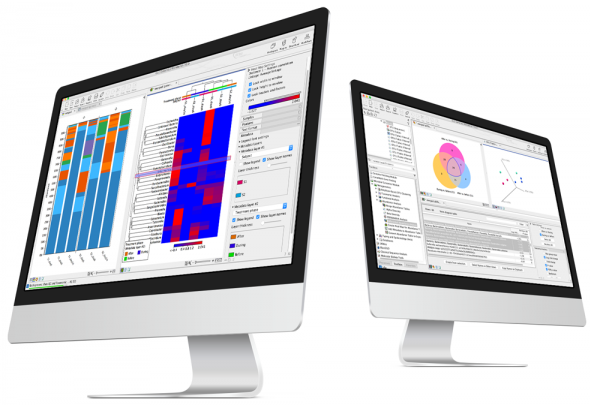
参考基因组数据库
1) 从NCBI广泛收集的微生物中直接导入细菌、古菌、病毒、真菌和原生动物参考基因组。
2) 一个表提供了涵盖的分类范围,装配状态和包含基因组的大小的概述。
3) 具有分类和组装质量的过滤器,用户可以优化数据库内容,删除低质量的数据集,以获得更高的可信度,通过减少数据库条目的冗余减少计算机资源和分析时间。
4) 下载之前监视设计期间的预计文件大小。
5) 从NCBI病原体检测项目NCBI RefSeq下载病原特异性参考基因组深度数据库。
Workflows
具有预先配置好的但也可自定义的微生物的生物信息分析流程,用户从原始的NGS reads 到完成交互式的视觉结果只需要几个步骤。
申请试用
微信公众号

