产品介绍
产品简介
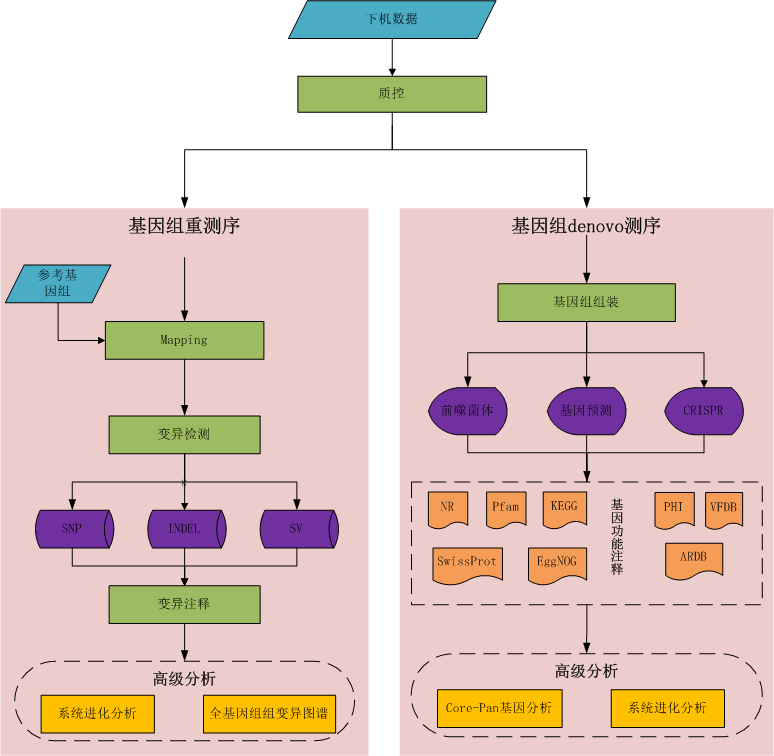
微生物全基因组测序是指对待研究的微生物进行全基因组测序,是获得未知生物的基因组或检测与已知生物基因组间的差异,以及比较多个样品基因组的重要手段,适用于微生物功能鉴定,差异检测及其他比较基因组学的研究。
全基因组重测序数据分析策略
1. 从测序所得到的原始数据出发,首先对原始数据进行质控,得到有效数据(Clean data);
2. 基于有效数据与参考基因组进行Reads Mapping比对;
3. 基于比对结果进行SNP、INDEL和SV的统计和注释;
4. 基于变异的统计结果绘制变异分布图和全基因组变异图谱;
5. 根据分析需求,可选择高级分析,深入挖掘物种的系统进化关系。
全基因组denovo测序数据分析策略
1. 从测序所得到的原始数据出发,首先对原始数据进行质控,得到有效数据(Clean data);
2. 基因组大小及杂合度评估,判断是否符合组装条件;
3. 基于有效数据,进行微生物基因组组装;
4. 基于组装结果,进行基因组组分分析:编码基因预测,前噬菌体及CRISPR预测;
5. 基于预测得到的基因序列,进行基因功能分析:包括通用基因功能注释(NR, GO, COG, KEGG, Pfam, SwissProt)和 病原细菌致病性和耐药性分析;
6. 根据分析需求,可选择高级分析,深入挖掘物种的系统进化关系。
数据分析内容
| 测序类别 | 分析类别 | 分析内容 |
全 基 因 组 重 测 序 |
与参考基因组比对情况分析 | 测序深度及覆盖度统计表 |
| 测序深度分布图 | ||
| 参考基因组各位置区间的深度分布图 | ||
| SNPs检测及注释 | SNPs位点类型及基因型 | |
| SNPs在参考基因组上的分布 | ||
| SNPs注释信息统计表 | ||
| INDEL检测及注释 | INDEL位点类型及基因型 | |
| INDEL在参考基因组上的分布 | ||
| INDEL注释信息统计表 | ||
| SV检测及注释 | SV变异类型 | |
| SV变异引起的染色体结构变化 | ||
| 高级分析 | 系统进化分析 | |
| 全基因组变异图谱 | ||
全 基 因 组 denovo 测 序 |
基因组组装 | 基因组杂合度评估 |
| 基因组频次深度分布图 | ||
| 组装结果统计 | ||
| 基因组组分分析 | 编码基因统计 | |
| 编码基因长度分布图 | ||
| 前噬菌体预测 | ||
| CRISPR位点预测 | ||
| 基因功能注释 | NR, SwissProt蛋白注释 | |
| KEGG,EggNOG,Pfam蛋白功能分类注释 | ||
| ARDB, PHI, VFDB致病和耐药性注释 | ||
| 比较基因组分析 | 共线性分析 | |
| 变异检测 | ||
| 高级分析 | Core-Pan基因分析 | |
| 系统进化分析 |
其他问题?
微信公众号

